Инженерный тур. 2 этап
В 2024 году Нобелевская премия по химии была присуждена за выдающиеся достижения в области предсказания структуры белков с помощью искусственного интеллекта. Эти исследования открыли новые горизонты в понимании молекулярных механизмов, лежащих в основе биологических процессов, и сделали инструменты ИИ, такие как AlphaFold, неотъемлемой частью современных биохимических исследований. Модели, подобные AlphaFold, уже помогли ускорить разработку новых лекарств, изучение заболеваний и создание катализаторов для химических реакций.
Задача будет состоять в том, чтобы использовать возможности AlphaFold для исследования структуры белка.
Дополнительные материалы: https://disk.yandex.ru/i/30pXFsv2exBsuQ.
Индивидуальные задачи второго этапа инженерного тура открыты для решения. Соревнование доступно на платформе Яндекс.Контест: https://contest.yandex.ru/contest/69897/enter/.
Получена аминокислотная последовательность белка, который участвует в клеточных сигнальных процессах. Задача состоит из двух частей:
Использование AlphaFold для предсказания структуры белка (30 баллов)
Предсказание трехмерной структуры белка.
- Необходимо использовать готовый код AlphaFold для получения трехмерной структуры белка по данной аминокислотной последовательности. Код предоставлен в виде ссылки.
- После успешного выполнения кода будет получен файл формата
.pdb(Protein Data Bank), который содержит пространственные координаты всех атомов белка.
Расчет числовой характеристики (70 баллов)
Расчет характеристик белка на основе его структуры.
- На основе полученной трехмерной структуры рассчитайте среднее расстояние между \(\alpha\)-углеродами (\(\ce{C\alpha}\)) аминокислот. Нужно вычислить среднее расстояние между атомами \(\ce{C\alpha}\) для каждой пары соседних аминокислот по всей цепи. В качестве ответа укажите это среднее расстояние с точностью до двух знаков после запятой.
Для обеих подзадач даны исходные данные:
Пример аминокислотной последовательности: MAAHKGAEHHHKAAEHHEQAAKHHHAAAEHHEKGEHEQAAHHADTAYAHHKHAEEHAAQAAKHDAEHHAPKPH.
Код на Python для использования AlphaFold:
Ссылка для загрузки: https://disk.yandex.ru/d/1nkhFbtIh8GliQ.
Блокнот Google Colab: https://colab.research.google.com/drive/19nFATJVWEo1ANfyBJB4M88Vug02rJZX5?usp=sharing.
Для решения этой задачи нужно выполнить следующие действия:
Запуск кода AlphaFold:
- Используйте предоставленный Google Colab-ноутбук.
- Следуйте инструкциям в ноутбуке для загрузки и выполнения кода.
Генерация PDB-файла:
- После успешного выполнения скрипта будет получен файл
.pdb, содержащий пространственные координаты атомов белка. - Предсказанная структура, которая должна быть получится можно скачать по ссылке: https://disk.yandex.ru/d/8cTUN0Vg_L1VaQ.
- После успешного выполнения скрипта будет получен файл
Анализ структуры файла:
- В файле
.pdbсодержится информация о положении атомов белка. - Пример структуры:
MODEL 1 ATOM 1 N MET A 1 22.283 -7.429 -15.904 1.00 47.82 N ATOM 2 CA MET A 1 21.229 -7.778 -14.956 1.00 47.82 C ATOM 3 C MET A 1 20.370 -6.563 -14.625 1.00 47.82 C ATOM 4 CB MET A 1 21.828 -8.360 -13.675 1.00 47.82 C ATOM 5 O MET A 1 20.162 -6.245 -13.453 1.00 47.82 O ATOM 6 CG MET A 1 22.473 -9.724 -13.864 1.00 47.82 C ATOM 7 SD MET A 1 22.051 -10.894 -12.515 1.00 47.82 S ... ATOM 572 CD2 HIS A 73 17.277 3.821 -35.831 1.00 49.34 C ATOM 573 ND1 HIS A 73 18.969 2.895 -36.859 1.00 49.34 N ATOM 574 CE1 HIS A 73 18.160 1.921 -36.476 1.00 49.34 C ATOM 575 NE2 HIS A 73 17.126 2.457 -35.851 1.00 49.34 N TER 576 HIS A 73 ENDMDL END
- В файле
Среднее расстояние между \(\alpha\)-углеродами или объем белка (в зависимости от выбранного задания): 3,77 Å.
Определение показателей IC50 (концентрация ингибитора, которая вызывает 50% ингибирование) и Ki (константа ингибирования) для белков является важной задачей в области биохимии, фармакологии и разработки лекарств. Эти показатели позволяют оценить эффективность потенциальных ингибиторов ферментов или других белков, что критично для разработки новых терапевтических препаратов.
Традиционно IC50 и Ki определяются с помощью экспериментальных методов, таких как ферментативные анализы. Однако такие эксперименты могут быть трудоемкими, дорогими и требовать значительных ресурсов. В связи с этим все большее внимание уделяется разработке методов машинного обучения для прогнозирования этих значений на основе известных структур белков и ингибиторов, а также их взаимодействий.
Командные задачи второго этапа инженерного тура открыты для решения. Соревнование доступно на платформе Яндекс.Контест: https://contest.yandex.ru/contest/69919/enter/.
Участникам предлагается собрать и проанализировать данные по белкам и их ингибиторам, включая значения IC50 (концентрация полумаксимального ингибирования) и Ki (константа ингибирования). Сведения можно получить из открытых баз данных (ChEMBL, PubChem, PDB) и научных статей. Подготовить набор данных с найденной информацией. После сбора данных участники должны построить регрессионные модели машинного обучения для предсказания IC50 и Ki на основе различных молекулярных признаков.
Язык программирования: Python 3.8 и выше.
Изучение и анализ IC50 и Ki
Найти и прочитать статьи о значении
IC50(концентрация полумаксимального ингибирования) иKi(константа ингибирования) в биохимии. Подготовить литературный обзор по теме, оформить в виде презентации.Примеры статей:
- Krippendorff B.-F., Neuhaus R., Lienau P., Reichel A., Huisinga W. Mechanism-Based Inhibition: Deriving KI and kinact Directly from Time-Dependent IC50 Values // Journal of Biomolecular Screening. 2009. Т. 14, № 8. С. 913 — 923. DOI: 10.1177/1087057109336751.
- He Y., Zhu Q., Chen M., Huang Q., Wang W., Li Q., Huang Y., Di W. The changing 50% inhibitory concentration (IC50) of cisplatin: a pilot study on the artifacts of the MTT assay and the precise measurement of density-dependent chemoresistance in ovarian cancer // Oncotarget. 2016. Т. 7, № 40. С. 66703 — 66713. DOI: 10.18632/oncotarget.12223.
- Kalliokoski T., Kramer C., Vulpetti A., Gedeck P. Comparability of Mixed IC50 Data — A Statistical Analysis // PLOS ONE. 2013. Т. 8, № 4. Ст. e61007. DOI: 10.1371/journal.pone.0061007.
- Burlingham B. T., Widlanski T. S. An Intuitive Look at the Relationship of Ki and IC50: A More General Use for the Dixon Plot // Journal of Chemical Education. 2003. Т. 80, № 2. С. 214 — 218. DOI: 10.1021/ed080p214.
Сбор данных
Использование ChEMBL, PubChem или PDB для получения данных о биологической активности соединений, а также любых других доступных открытых баз данных. В качестве альтернативного метода допускается применение методов автоматизации для сбора (например, API или скраппинг), а также поиск информации в научной литературе, в том числе в научных статьях.
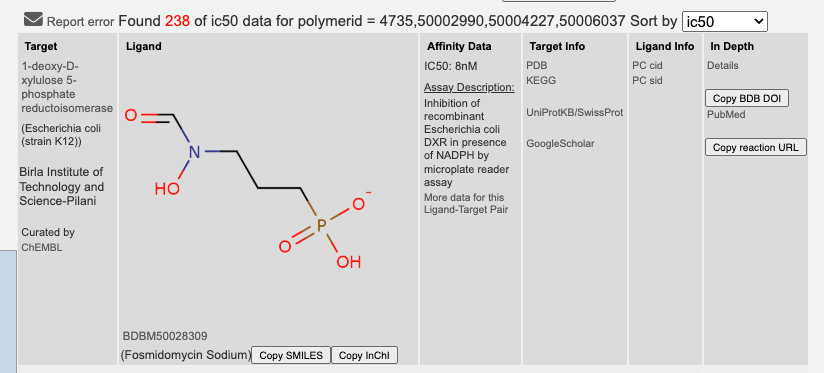
Рис. 2.1.Данные, полученные из научных статей, будут оцениваться выше при оценке этапа сбора информации. Это связано с тем, что статьи представляют собой первоисточники, содержащие тщательно проверенные результаты экспериментов и детализированные описания условий их проведения.
Пример: можно собрать данные с ресурса https://www.bindingdb.org/rwd/bind/ByKI.jsp?specified=IC50 (рис. 2.1).
Это открытая база данных с возможностью скачивания датасетов.
Предобработка данных
Формирование итоговой базы данных, представляющей собой двухмерную таблицу, содержащую следующие столбцы: SMILES-нотации малых молекул, которые связываются с рассматриваемым белком; значение константы
IC50, константыKi, ссылки на источник, откуда было взято то или иное значение константы (отдельный столбец для каждой константы). Провести канонизацию SMILES для молекул, удалить дубликаты молекул. Необходимо сохранить полученный набор данных в формате.csv. Пример итогового преобразованного набора данных приведен в таблице 1.1.Canonical Smiles Ki Link_Ki IC50 Link_IC50 S=P(N1CC1)(N1CC1)N1CC1 4.23 PDB 4.23 PDB CC1CCC2C(C)C(=O)OC3OC4(C)CCC1C32OO4 2.25 ChemBL 2.25 ChemBL COc1cc(CNC(=O)CCCCC=CC(C)C)ccc1O 4.37 PubChem 4.37 PubChem Извлечение признаков
Для построения модели машинного обучения требуется формирование датасета с различными дескрипторами и фингерпринтами молекул. Также можно использовать предобученные трансформеры для получения эмбеддингов молекул. Ниже приведены возможные инструменты для получения необходимых признаков:
- Фингерпринты (например, Morgan, MACCS).
- Дескрипторы библиотек (RDKit, Mordred).
- Эмбеддинги трансформеров (СhemBERT).
Обучение регрессионных моделей
Участникам необходимо применить модели машинного обучения, такие как
Пример кода для обучения модели:CatBoost,XGBoost, иLightGBM, для предсказания значенийIC50иKiна основе молекулярных признаков, собранных на предыдущих этапах. Эти модели основаны на градиентном бустинге деревьев решений, что делает их эффективными для задач с неструктурированными и сложными данными, такими как молекулярные дескрипторы и фингерпринты.Pythonfrom sklearn.ensemble import GradientBoostingRegressor reg = GradientBoostingRegressor(random_state=0) reg.fit(X_train, y_train) GradientBoostingRegressor(random_state=0)Разделение выборки. Необходимо разделить данные на тренировочную и тестовую выборки. Рекомендуется использовать соотношение 80/20, чтобы выделить достаточно данных для обучения модели и ее последующего тестирования.
Важным аспектом является использование
Пример разделения выборки:random seedдля разделения данных. Это обеспечит воспроизводимость результатов, что необходимо для корректного сравнения моделей. Например, можно использоватьrandom_state=42или любой другой фиксированныйseedдля консистентности.Pythonfrom sklearn.model_selection import train_test_split X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, shuffle=True, random_state=42)Обучение моделей. Применить модели
CatBoost,XGBoost, иLightGBMк тренировочным данным, используя соответствующие фреймворки.Оценка качества модели. Оценить каждую модель на тестовой выборке с использованием метрик
Пример оценки точности модели по трем метрикам:MAE,RMSE, иR\(^2\) (доступна реализация через библиотекуscikit-learn), сравнить результаты моделей и выбрать наилучшую, основываясь на точности предсказания.Pythonfrom sklearn.metrics import mean_absolute_error, mean_squared_error, r2_score y_ki_pred = model.predict(X_test) mae = mean_absolute_error(y_ki_test, y_ki_pred) rmse = mean_squared_error(y_ki_test, y_ki_pred, squared=False) r2 = r2_score(y_ki_test, y_ki_pred)Заключительный анализ
Представить результаты в виде отчета или презентации. Интерпретация полученных моделей и выводы о взаимодействиях белков с ингибиторами, а также вывод о применимости тех или иных методов генерации признаков.
Ответ к задаче должен строго содержать zip-архив, содержащий:
- презентацию, содержащую литературный обзор и описание хода выполнения задания.
.pyили.ipynb, включающий в себя код для решения задачи.- преобразованный набор данных в
.csvформате.
Пример отчета: https://disk.yandex.ru/i/09GEADjD5bPCpg.
Постановка задачи и теоретическое обоснование (15%):
- Полнота изучения значения
IC50иKi. - Корректный выбор белка и описание методов сбора данных.
- Полнота изучения значения
Сбор и обработка данных (25%):
- Оценка правильности сбора данных (необходимые ссылки на источники, полнота данных, соответствие запрашиваемому формату таблицы).
- Качество очистки и предобработки данных, канонизация SMILES.
Полный балл возможен только в случае полной очистки данных от дубликатов и пропусков. Без канонизации SMILES задание оценивается в 0 баллов. В случае непредставления базы данных в отчете, задание также оценивается в 0 баллов.
Извлечение признаков и создание датасета (30%):
- Опробованы разнообразные признаки (фингерпринты, дескрипторы, эмбеддинги). Необходимо использовать минимум три разных вида признаков для обучения. Например, два вида фингерпринтов и дескрипторы RDKit.
- Осуществлен отбор признаков с помощью корреляционного анализа.
Обучение и оценка моделей (20%):
Проведена оценка точности обученных моделей, выведены все запрашиваемые метрики.
R\(^2\) модели на тестовых данных должен быть больше 0,5.Оформление и интерпретация результатов (10%):
Логичность представления данных, выводов и рекомендаций.